Plumed-02-PATHMSD-ABMD
在复杂的反应或者构象变化中,通常需要大规模的构象采样,甚至是增强采样来加速分子动力学模拟。而这些复杂的构象变化过程往往难以通过单一的cv进行描述,比如激酶激活过程中activation loop的 open-close transiton (eg. CDK2 2c5x -> 2c5y) 。Plumed中的PATHMSD这个cv或许会有所帮助。 # PATHMSD 使用PATHMSD作为cv,需要预先定义反应路径,通常可以通过Gromacs的插值,或者弹性网,或者Plumed的pathtool,或者GISMO插件,或者Plumed-GUI的 Structure->Build reference structure 等工具产生初猜的路径(尽量使这个路径足够均匀)。 确定了初猜的反应路径之后,便可得到反应路径上的N个构象(包含两个端点),然后反应的过程就可以通过如下公式进行计算: \[ S(X)=\frac{\sum_{i=1}^{N} i\ \exp^{-\lambda \vert X-X_i \vert }}{ \sum_{i=1}^{N} \exp^{-\lambda \vert X-X_i \vert } } \] 其中\(\vert X-X_i \vert\)表示待分析的构象\(X\)与路径上的某个构象\(X_i\)之间的距离,\(\lambda\) 为一个正数(下文会具体说明)。 由以上公式可以看出,当\(X\)接近构象\(X_i\)时,\(\vert X-X_i \vert\)近似于0,对应的\(\exp\)指数函数的值接近于1,反之若\(X\)距离\(X_i\)较远,则得到的指数函数的值就接近于0.所以,通过调节\(\lambda\)的值,可以做到当\(X=X_j\)时,\(S(X)=j\). \(\lambda\)的值通常由如下公式获得: \[ \lambda=\frac{2.3 (N-1) }{\sum_{i=1}^{N-1} \vert X_i-X_{i+1} \vert } \]
其中\(\vert X_i -X_{i+1}\vert\)表示路径上连续两帧之间的距离(通常是两个构象之间的MSD(即RMSD的平方)),最好可以使预先定义的路径上每两帧之间的距离比较接近。
1 | p1: PATHMSD REFERENCE=all.pdb LAMBDA=50.0 |
在参考构象的PDB文件中包含多个构象(X1,X2,...,XN),构象之间使用END隔开。不需要在此文件中放入体系里所有的原子,只需要放入你认为需要的那一部分即可,比如你可以删掉溶剂,离子甚至是一个结构域。
此外,需要注意\(\lambda\)的单位,在Plumed中默认的长度单位是纳米。
计算PATHMSD之后产生两个参数,sss是当前构象沿着反应路径的过程,zzz是这个构象和反应路径上与它最接近的构象之间的距离Z(X)。
\[
Z(X)=-\frac{1}{\lambda}\log (\sum_{i=1}^{N} \ \exp^{-\lambda \vert X-X_i
\vert })
\] 如果\(\lambda\)选择的合理,则当\(X=X_i\)时,上式的结果即为 \(\vert X-X_i \vert\)
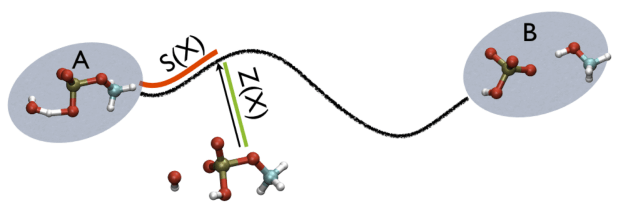
从下图中可以看出,这两个参数描述了某个构象在反应路径上所处的位置:
1 | p1: PATHMSD REFERENCE=all.pdb LAMBDA=50.0 NEIGH_STRIDE=4 NEIGH_SIZE=8 |
如果在每一步计算中都拿路径上所有的构象来计算S(X)速度会降低,所以可以通过NEIGH_STRIDE=4和
NEIGH_SIZE=8来设置成每次计算S(X)时只使用反应路径上与X最接近的8个构象,并且每4步对这8个构象构成的列表进行更新。
reference PDB:
这个PDB文件中包含预先定义的路径上的每一个构象,构象之间使用END隔开,不需要将整个系统的所有原子都放入。
PDB文件的occupancy和bfactors这两列,前者的数值设为1表示使用对应的原子进行align,后者的数值设为1则表示使用对应的原子计算距离。所以,可以选定一部分原子用来align,另一部分原子进行距离计算。
ABMD
Case1
1 | cat > plumed.dat |
Case2
1 | cat > plumed.dat |