蛋白质子化工具ProteinPrepare
在进行分子模拟之前一般需要对蛋白的结构进行优化,包括确定质子化状态和优化氢键网络等。PlayMolecule平台的ProteinPrepare所提供的交互式和命令行工具(需下载离线版本)能够实现这一功能。
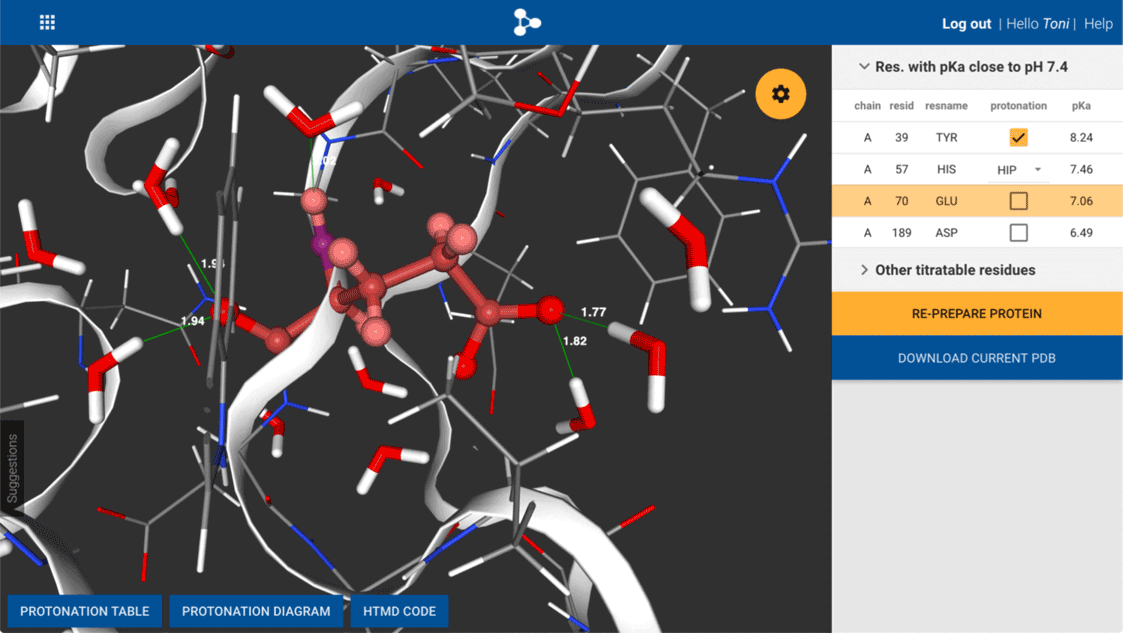 首先,用户上传pdb结构文件,然后选定需要处理的蛋白链,是否移除晶体水分子,是否将非标准氨基酸修改为标准氨基酸,并确定需要计算的pH值。
首先,用户上传pdb结构文件,然后选定需要处理的蛋白链,是否移除晶体水分子,是否将非标准氨基酸修改为标准氨基酸,并确定需要计算的pH值。
然后服务器后台会使用PROPKA-3.1来计算蛋白侧链的pKa值,然后通过如下公式计算质子化的概率p: \[ p=\dfrac {1} {1+10^{pK_{a}-pH}} \] 由上式可知,当\(\left| pH-pK_{a}\right| < 1\)时,概率p在0.1 - 0.9之间,这些残基的质子化状态并不是特别确定,因此ProteinPrepare会将这些残基列出,由用户自行决定质子化状态,当然,其它残基的质子化状态也可以由用户自行确定。
此后,服务器后台会使用PDB2PQR-2.1对氢键进行优化,主要包括补全缺失的重原子,然后加氢,然后对蛋白侧链进行优化(possibly flipping Gln, Asn, and His residues)。如果结构中含有水分子,那么蛋白-水分子,水分子-水分子的相互作用也会在优化的过程中被考虑。
计算完成后,ProteinPrepare会在主窗口的下方显示Protonation Table,
Protonation Diagram,
HTMD Code三个标签,分别对应一个包含所有信息的excel表格,一张标记残基pKa值的图片,以及对应处理过程的Python代码(用于离线版的命令行工具)。
主窗口的右方,会列出需要用户自行指定质子化状态的残基,以及所有其它可指定的残基。以上流程中,已经进行了第一次计算,如果对此结果满意,可直接下载目前已经处理好的pdb结构;若需要对某些残基的质子化状态进行手动指定,则指定完成后需点击Re-Prepare-Protein按钮再次进行计算。